pprofをWeb上で見られるツールを作って、ISUCON13に挑戦しました
デプロイ周りで手戻りが起こってしまい若干後悔が残る結果になりましたが、去年よりは着実にスコアを上げられた気がします。
最終スコアは33,789点で、694チーム中56位でした。
僕(シバニャン)は、メトリクス、オンメモリキャッシュ、NGワード登録時の判定、N+1の解消(ちょっとだけ)を担当しました。
学び
やるべきだったこと
- 大きな変更を入れるのではなく、じわじわと効きそうなものを入れていくべきだった。
- デプロイスクリプトのミスのチェックのために、コミットログが新しいか、ビルドが更新されているかを毎回確認するべきだった。
やってよかったこと
ボトルネックの特定に、felixge/fgprofとshiba6v/go-fprof、pprofpageが役に立ちました。
felixge/fgprof
fgprofは、pprofと似たように使用できますが、CPUを使っていない時間(DB待ちなど)も測定してくれます。
shiba6v/go-fprof
shiba6v/go-fprofは、去年のISUCONから導入しているツールです。 alpと比べると、privateメソッドや関数の途中などの好きな場所に計測を増やしていける点がメリットで、エラーの回数やbodyサイズを計測できないのがデメリットです。
計測すると、以下のような結果が得られます。
FProf Result [us] Sum 1172, Max 1172, Avg 1172, Min 1172, Count 1, main.GetPaymentResult:L15 Sum 6122746, Max 224235, Avg 21333, Min 661, Count 287, main.getTagHandler:L27 Sum 103671832, Max 456753, Avg 73526, Min 658, Count 1410, main.getReactionsHandler:L38 Sum 387096, Max 108662, Avg 21505, Min 960, Count 18, main.getStreamerThemeHandler:L60 Sum 31469279, Max 3999090, Avg 1656277, Min 414043, Count 19, main.getUserStatisticsHandler:L64 Sum 81552906, Max 253907, Avg 59397, Min 1459, Count 1373, main.getLivecommentsHandler:L71 Sum 36140357, Max 340977, Avg 122095, Min 340, Count 296, main.reserveLivestreamHandler:L72 Sum 31210139, Max 3987903, Avg 1642638, Min 407561, Count 19, main.getUserStatisticsHandler:L81 ...
pprofpage
pprofpageは、今年新しく作ったツールです。pprofやfgprofの計測結果をPOSTすると、URLを発行してくれます。

PPROF_PATH=$(curl -X POST -F file=@/home/isucon/tmp/cpu.pprof https://emaaxnj6hk.execute-api.ap-northeast-1.amazonaws.com/prod/pprof/register)
echo https://emaaxnj6hk.execute-api.ap-northeast-1.amazonaws.com/prod${PPROF_PATH}
ファイルをURLに向けてpostすればページが完成します。 LambdaとS3、API Gatewayでサーバーレスで作ったので維持費がほぼ0円で、スパイクが来ても耐えられるはずです。(DDoSされた場合を除く)
ざっくりやったこと
やったこと
- インデックスを貼りまくる
- N+1を解消
- userStatistics
- reactions
- オンメモリキャッシュ
- User
- Icon
- userのidで取る部分とnameで取る部分があるのでそれぞれ
- Livestream & Tags
- DBを分ける
- DB1台とApp1台になりました。
- NGワード登録時のNGワード判定をDBからAppに移す
やろうとしたけどできなかったこと
- DNSサーバーとAppを分けようとした
- totalTipsのN+1を分ける部分で時間切れ
時系列
序盤イケイケ期
10:24 4,134点
序盤は、メトリクスの整備を行い、LiveStreamとTagsのローカルキャッシュを入れ、DBを分けました。
12:00 7,005点
12:42 13,068点
中盤絶望期
13:42
立て続けにベンチがfailし、そこからinitializeが叩けなくなりました。
15:00
原因を切り分けていった結果、昼ごろからずっとコードが反映されていないことが発覚しました。
これまでにマージしたものは、ベンチが通ったと勘違いしていただけということが分かり、絶望の瞬間が訪れました。
15:30ごろに、デプロイスクリプトの修正が終わりました。
後半マージ期
15:30からは、ベンチが通らない状態だったmasterブランチを捨て、 最後のベンチが通る状態から、12:00-13:40の間の変更を確認しながらマージしていきました。 インデックスを貼り、User, Iconのキャッシュを入れ、userStatistics, reactionsのN+1を解消し、NGワード判定をDBからAppに移しました。
16:25 21,521点
16:51 25,835点
17:31 28,088点
最終チェック
競技終了時点でも、まだまだやれそうなことがありそうな気配がしていましたが、万全を期すために17:30ごろに終わりにすることにしました。
goccy/go-jsonへの切り替え、MySQLのconfig変更、ログ切り、再起動試験、点数ガチャを行いました。
17:49 33,789点
今年も楽しい問題でした。 運営の方々、ありがとうございました。 非同期周りのコーディングの訓練や、DBの知識の習得に役立っており、感謝しています。
部屋探しにおける高速インターネット回線の調べ方
部屋探しをする時に、その部屋に通っているインターネット回線が高速かどうかを調べる方法を備忘録としてまとめました。
- 要約
- 部屋探し時に、光回線3社のエリア確認ページでネット回線の有無を調べる。
- 出てきたプランから回線速度を確認する。
物件選びのときに、高速インターネット回線を確認しよう!
リモートワークには高速な光インターネット回線が重要です。既にアパートに光回線が導入されている場合は、共有部分から部屋までの工事だけで済むため、安価で済みます。既に高速な回線が導入されていなければ、大家と交渉してアパートに回線を引いてくる必要がありますが、これは時間もかかりますし、必ず出来るとは限りません。
そのため、物件選びの際に光回線がアパートまで来ているか確認することが大事です。
遅い光回線もある
光回線でも、アパート内の配線方式によっては通信速度が遅い場合があります。 これから紹介するエリア確認ページでは、その配線方式も調べることが出来ます。
光回線の有無は、エリア確認で調べる。
賃貸物件の高速インターネット回線の有無を調べるには、各回線会社のエリア確認ページが使えます。携帯電話番号の入力や、居住している必要はありません。(念の為、内見時にも光コンセントがあることを確認しましょう。)
3社調べればOK!
以下は、フレッツ光、auひかり、NURO光の3社のエリア確認サイトのURLと、その結果が表す下り速度です。
光コラボ(ドコモ光、Softbank光、OCN光、ビッグローブ光、楽天光、IIJmio光、excite MEC光など)は、フレッツ光と同じNTTの回線を使用しているので、光コラボを使いたい場合も、フレッツ光回線が通っていることを確認すれば十分です。 (関西だと、eo光 も選択肢に入ります。)
フレッツ光
まず、物件の住所を入力します。
- 物件の住所は、SUUMOで分からなくてもHOME'Sで分かる場合があります。
- HOME'Sでも住所が分からない場合は、ざっくりした住所+築年数を使って頑張ってGoogleで探しましょう。
- フレッツ光のエリア確認の住所の番地は、適当に入れてもアパートが出てくる場合があります。
- 部屋番号は適当に入れましょう。だいたいどの部屋でも同じです。
この画面が出たら、適当に押して先に進みましょう。

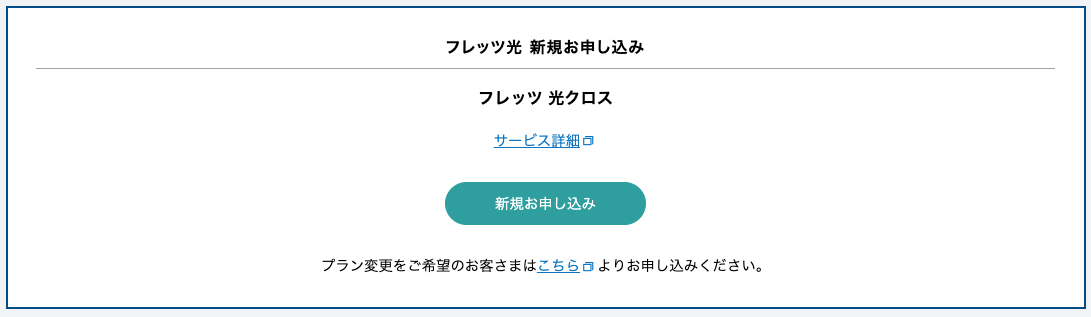
結果の画面は、東日本エリアでは大きく以下の2種類が確認できました。
1 回線が通っている
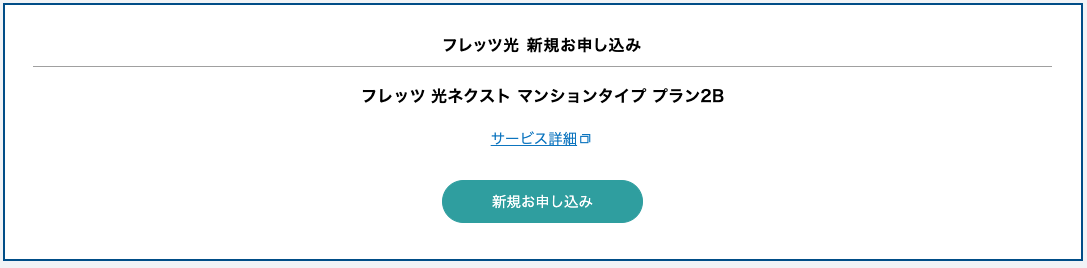
2 要確認(未導入の可能性が高い)

回線タイプの結果と最大速度の対応は、以下のとおりです。
- 結果の見方
参考: https://www.isdn-info.co.jp/order/202202_05.pdf
2の要確認の場合は、新築に近い場合は通っている場合もありますが、そうでない場合は通っていない可能性が高いです。 不動産屋に直接聞く必要があります。
物件の内見時には、フレッツ光回線が通っていることだけを確認すれば十分です。内見時には、回線が引いてあることの最終確認として光コンセントの有無を確認しましょう。念の為写真を撮っておくと良いです。
フレッツ光の場合は、NTT東/西日本以外の回線事業者(光コラボ)も選べます。事業者の選択は、入居契約後で大丈夫です。 光コラボに関しても、後で記事を書きたいです。
auひかり
フレッツ光と同様に、住所を検索します。

- 結果の見方
- 高速 (1Gbps以上、光配線方式)
- ギガ
- ホームタイプ (1ギガ、5ギガ、10ギガ)
- マンションミニギガタイプ
- 中速 (664Mbps、G.fast方式)
- タイプG (G契約) *タイプGの新規申し込みはG契約。
- 都市機構G (DX-G)
- 低速 (100Mbps以下、VDSL方式、LAN配線方式など)
- タイプG (V契約) *マンションタイプVから移行した場合。
- タイプV
- 都市機構
- 都市機構G (DX、16M (B)、16M (R))
- タイプE
- タイプF
- 高速 (1Gbps以上、光配線方式)
参考: https://www.soumu.go.jp/main_content/000725528.pdf
auひかりやNURO光は、既に通っているマンションはそこまで多くありません。 時間が無いときは、フレッツ光だけでも良いでしょう。
NURO光
フレッツ光と同様に、住所を検索します。
nuro導入済みだと分かっているマンションの住所を入れてみると、 crew/ が入ったURLが出てきました。
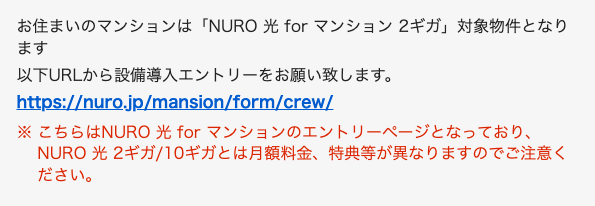
未導入の可能性が高いマンションの住所を入れてみると、crew/ の入っていないURLが出てきました。
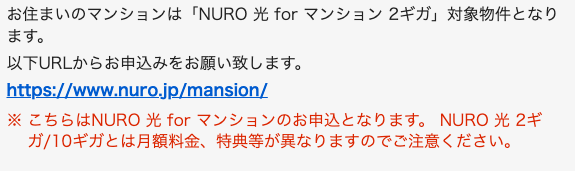
リンクをクリックして情報を入力すると、月額料金が出てきます。 未導入の場合でも、高い初期費用を払えば回線を引くことはできる可能性があります。
- 結果の見方
- 高速 (2Gbps以上、光配線方式)
- 10ギガ
- 2ギガ
- forマンション
- 高速 (2Gbps以上、光配線方式)
J:COM
J:COMのインターネットは、ケーブルテレビ回線を使っています。物件情報に「インターネット(CATV)」と書いている場合は、J:COMのインターネット回線が付いています。 残念ながら、J:COMの回線はアップロードが非常に遅いのでおすすめしません1。アップロード速度が重要ではないYouTubeなどは問題なくても、オンラインゲームやZoomが不安定になります。
J:COMでも高速なケースはありますが、それは「J:COM 光 NET 1Gコース」というauひかり回線をJ:COMが提供しているものなのでこれはJ:COMのケーブルテレビ回線とは異なるものです。
まとめ
初見では意味不明な物件のインターネット回線は、実は住む前に調べてある程度分かるということです。 物件探しをしている方は、頑張ってください。
免責
情報の正確性は保証できず責任を取れないので、正しい情報を知りたい場合は公式サイトをあたってください。 このブログを信じてショボい回線の家に住んでしまっても許してください。
ブログっぽい雑感
本当は、賃貸検索サイトのChrome拡張や、住所を入れると各回線の状況が一度分かるツールを作りたかったのですが、住所やスクレイピングの扱いが面倒でやめました。各社のプランが非常にわかりづらい点を課題に感じていたので、このまとめである程度役割は果たせると思います。
auひかりは同じタイプなのに回線速度が違うものがあるのが分かりづらいですね。フレッツ光は名前がインフレしていっているので、最強無敵スーパーウルトラ神神神チーム2 のような印象を受けました。
追記
この話に関連するインタビューが記事になりました! 引越しインターネット回線で失敗しないポイント|引越し見積もり・比較【SUUMO】
Protobufの破壊的変更を検知するProto Breaking Change Detectorを使ってみた
概要
Protobufの破壊的変更を検知するProto Breaking Change Detectorというツールを使ってみたら、良さそうだった。 github.com
経緯
uber/protool を使うと破壊的変更を検知できるようだったが、リポジトリがarchiveされている。
prototoolの代替として示されているBufの破壊的変更検出 は、buf.yamlをディレクトリに置くというBufのルールに従わなければ利用できない。(たぶん。やり方を知っている人がいたら教えて欲しい。)
方法
動かしてみる
mainブランチと現在のワークツリーで、破壊的変更が発生していないかチェックする。
Proto Breaking Change Detector をインストールする。
pip install git+https://github.com/googleapis/proto-breaking-change-detector.git
2つのディレクトリ間でのprotobufの破壊的変更を検知できる。
# --original_api_definition_dirs: 比較元の--proto-pathに指定するディレクトリをカンマ区切りで
# --original_proto_files: 比較元のbeaking changeを検出したいファイルをカンマ区切りで
# --update_api_definition_dirs: 比較先の--proto-pathに指定するディレクトリをカンマ区切りで
# --update_proto_files: 比較先のbeaking changeを検出したいファイルをカンマ区切りで
PROTOCOL_BUFFERS_PYTHON_IMPLEMENTATION=python proto-breaking-change-detector \
--original_api_definition_dirs=${ORIGINAL_PROTO_PATH} \
--update_api_definition_dirs=${UPDATE_PROTO_PATH} \
--original_proto_files=${ORIGINAL_FILE} \
--update_proto_files=${UPDATE_FILE} \
--human_readable_message
実行すると、detected_breaking_changes.jsonというファイルが作られて、そこに非破壊的変更("change_type": "MINOR")や、破壊的変更("change_type": "MAJOR")を含む変更の情報が入る。
mainブランチからの破壊的変更を検知してみる
mainブランチと今いるブランチの間で、破壊的変更が無いか比較してみるために、サンプルコードを作った。 github.com
cloneしてブランチを切る。
git clone git@github.com:shiba6v/pbcd_trial.git cd pbcd_trial git checkout -b feature/hoge
元のmainブランチのproto/fuga/fuga.proto は、次のような定義にした。
syntax = "proto3";
import "hoge.proto";
message Fuga {
int64 id = 1;
Hoge hoge = 2;
}
mainブランチをcloneしてきたままの状態で、proto-breaking-change-detectorを実行するスクリプトを実行する。
sh detect.sh
もちろんoriginのmainブランチから変更はないので、detected_breaking_changes.jsonの中身は空になっている。
[]
次に、破壊的変更と非破壊的変更を加えてみる。
具体的にはHoge hoge = 2;をstring hoge = 2;に変更して、int64 count = 3;を追加する。
syntax = "proto3";
import "hoge.proto";
message Fuga {
int64 id = 1;
string hoge = 2;
int64 count = 3;
}
再度sh detect.shすると、
$ sh detect.sh pbcd_trial/protobuf/src: warning: directory does not exist. pbcd_trial/protobuf/src: warning: directory does not exist. fuga.proto:3:1: warning: Import hoge.proto is unused. fuga.proto L7: The type of an existing field `hoge` is changed from `message` to `string` in message `..Fuga`.
mainブランチから見ると破壊的変更("change_type": "MAJOR")が起こっていることが確認できる。
[
{
"category": "FIELD_ADDITION",
"location": {
"proto_file_name": "fuga.proto",
"source_code_line": 8
},
"change_type": "MINOR",
"extra_info": null,
"subject": "count",
"oldsubject": "",
"context": "..Fuga",
"type": "",
"oldtype": ""
},
{
"category": "FIELD_TYPE_CHANGE",
"location": {
"proto_file_name": "fuga.proto",
"source_code_line": 7
},
"change_type": "MAJOR",
"extra_info": [
"message Fuga {",
"hoge"
],
"subject": "hoge",
"oldsubject": "",
"context": "..Fuga",
"type": "string",
"oldtype": "message"
}
]
フィールドの追加は、非破壊的変更("change_type": "MINOR")と判定されており、これは正しい。
今後知りたいこと
- ファイルが追加されるときや、messageがファイルを移動したときはどう検知されるか?
protobuf/src: warning: directory does not exist.のWarningを抑えたい。protobuf/srcがデフォルトで--proto-pathに追加されている。
結論
を使おう。
ISUCON12予選に参加した
ISUCON12予選に参加しました。 最終ベンチ結果は6815点で、最高ベンチ結果は10625点、failの可能性は高いです。 もし結果発表でfailしていたらブログを書く気に多分ならないと思う(あと記憶力が弱い)ので、今のうちに当日やったことを中心に振り返っていきます。
追記 (2022/7/25): failしていました :cry:
追記 (2022/11/26): プロファイラ的なツールを公開しました。 github.com
自分がやったこと
ファイルロックがボトルネックになっているっぽいことを見つけた
alpのログを見ると、ランキングやプレイヤー詳細のAPIがボトルネックになっていることが分かりました。

alpだとエンドポイント単位でしか見られないんですが、pprofはポートを立ててWeb UIから見なければ詳しくは見られない上にちょっと見づらい・・・ということで、このときから作っていたツール1 を使って、関数ごとにかかった時間を計測しました。
ISUCON用ツールを作ってたらISUCONの練習をする時間がなくなりそう
— シバニャン (@_6v_) 2022年7月16日
alpで一番重かったランキングAPI (competitionRankingHandler) を分割して見たところ、一番時間がかかっているのはSQL文ではなく、ファイルロックをしている部分(competitionRankingHandler 4)でした。

ファイルロックをする関数(flockByTenantID)も、時間がかかっていることが分かります。
![]()
ファイルロックを消した (failの戦犯)
SQLiteのロック適当に外したらたまにfailするようになってずっと泣いていた #isucon
— シバニャン (@_6v_) 2022年7月23日
お題のアプリケーションはマルチテナントSaaSで、管理側はMySQLを使っており、テナントに入っている側はそれぞれ別のSQLiteを使っていました。
ロックをかけている部分のコードには、
// player_scoreを読んでいるときに更新が走ると不整合が起こるのでロックを取得する
とコメントがあるのですが、DBの読み込み時にかかっている排他ロックが、読み込みが非常に遅い原因になっていました。
読み込み時には共有ロックをかけると読み込みが同時に行えて性能が上がるだろうということで、ファイルロックをやめてSQLiteのトランザクションを使おうとしました。
トランザクションに書き換えると、error retrievePlayer: error Select player: id=395f19764, database is locked というエラーが出てしまうものの、スコアは5000点から10000点近くまで上昇しました。
追記 (2022/7/25): 正しくトランザクションが貼れていてもこのエラーは出るので無視してよかったらしい。そんな・・・
いろいろ試行錯誤した結果、このエラーが出たり出なかったりしてスコアが0になったり10000になったりしました。これが失格の原因になるのは困るということで、⚠️ロックを一切かけない⚠️ことにしました。このあたりで既に完全に沼に入っているので、冷静な判断ができていればrevertして5000点から再開していたと思います。失格になったらこの変更のせいなのでごめんなさい(泣)
テーブル単位で徐々にSQLiteからMySQLに移していってるのすごいなあhttps://t.co/8JcnHWHn1m
— シバニャン (@_6v_) 2022年7月23日
リーダーボードで上位だった方のコミット履歴を見ると、SQLiteからMySQLにテーブル単位で移していました。 SQLiteのロックを外すことを考えるよりも先にその戦略を取れば、普段使っているMySQLのトランザクションが使えるのでやりやすかったと思います。 SQLiteからMySQLへのリプレースは弊チームではリスクが高いと判断して取り組まなかったことを考えると、部分的に置き換えるというのはやはりISUCONにおいて重要戦略であると思いました。
追記 (2022/7/24): ファイルロックをsync.RWLockとsync.RLockにする案は思いついたんですが、tenant IDごとにロックを分けるためにmapを作る必要があって、そのmapを管理するためにロックが必要で・・・となって面倒くさそうでやめてしまいました。tenant IDは固定でmapはreadだけなので、本当はそんなに難しくなかっただろうなと思います・・・こっちでやればよかった:cry:
GoDocを誤読
— シバニャン (@_6v_) 2022年7月24日
ファイルロックをRLockにもしてみたんですが、failしたのでロックが取れるまで待つ実装は自前でやらなければいけないのかなと勘違いしました。It will wait until it is able to obtain the shared file lock. とあるので、その方針でも自信をもってやればできただろうと思います。
https://pkg.go.dev/github.com/gofrs/flock#Flock.RLock
チームメンバーがやったこと
IDジェネレータをUUIDにした (AokabiC)
スロークエリログを見ると、id_generatorという謎テーブルへのINSERTが一番遅いことが分かりました。被らない連番IDを取るために、MySQLにわざわざレコードを追加して採番していたので、これをUUIDに変更してくれました。
REPLACE INTO id_generator (stub) VALUES ('S')
AokabiC氏の目の付け所はいつも洗練されているのですごいなあと思います。
App, DBの2台構成にした (zatton)
ISUCON12予選では3台のサーバーが与えられますが、AppとDBを分離するのは、 - htopをするとAppとDBのどちらがボトルネックになっているかわかる - 負荷が軽くなる の2点のメリットがあるため、早い段階で分離しました。
弊チームは、/etcなどの設定ファイルは権限周りが面倒なのでGit管理せずにzatton氏がVimでゴリゴリ直接書いていく方針で、zatton氏がサクッとやってくれました。
visit_historyへの書き込みを減らす (AokabiC)
visit_historyはランキング取得のタイミングで履歴を書き込むのですが、このテーブルは、最新のものしか取得されていません。そこで、visit_history_summerizeというテーブルを作って、最新のものだけを書き込むようにしました。インデックスも貼りました。
自分がやろうとしたこと
player_score を剥がす

ファイルロックがボトルネックから外れた後は、player_scoreテーブルを使う関数がボトルネックになってきたのでこれをRedisに移そうとしました。しかし、整合性チェックでfailしてデバッグする時間もなく断念。RedisではなくMySQLにInsertしている方が整合性に関して安心できるという点で良い方針だったのかもしれません。
チームメンバーがやろうとしたこと
3台構成にする (zatton)
3台構成にする際は、Appを2台(nginx+AppとApp)にするかDBを2台(リードレプリカや垂直分割)にするか、その他の構成にするかが悩みどころです。 今回は、2台構成にしたところAppのCPU負荷が100%に張り付いていたので、Appを2台にすることを考えました。しかし、Appサーバーのファイルシステム上にはSQLiteがあり、Appを2台にするならNFSなどで同期しなければいけないことが分かります。ということで、これは一旦放置することにしました。
player_scoreへの書き込みを減らす (AokabiC)
player_scoreもvisit_historyと同様に書き込みが減らせそうということで、取り組んでいましたが時間が足りず断念。
感想
既存のISUCON攻略法が使えない感じになっていてすごい問題だった #isucon
— シバニャン (@_6v_) 2022年7月23日
既存のISUCON攻略法を封じてくるような問題だったので、面白かったです。 具体的には、DBがSQLiteとMySQLに分離されていたので、MySQLのスロークエリログには出ないボトルネックがありました。
余談
当日は、SQLiteやMySQLがトレンド入りして盛り上がりました。

ISUCON界隈の外からはMySQLトレンド入りの原因が謎だったっぽい。
MySQLがトレンドに入っていて、
— 大和(で)哲 (@deyamato) 2022年7月23日
「なぜトレンドに? 」と思ってトレンドを見ると
大量の「なぜMySQLがトレンドに?」で構成されていた。
なんという再帰的な……。
— シバニャン (@_6v_) 2022年7月23日
ここは違うよ、というのがあれば、TwitterのDMで教えてもらえると助かります。
セマンティックセグメンテーションでやっていく年賀状作成 2022
この記事は、CAMPHOR- Advent Calendar 2021 の3日目の記事です。
概要
年賀状やメッセージカード作成に役立つWebツール、SSフォトカードメーカー を作りました。
このツールを使うと、写真からこういった画像を作成できます[^1]。

全面写真の年賀状と「上に文字が乗らない問題」
年末といえば、年賀状作成ですね。最近はSNSで送る人もいるみたいです。 テンプレートに小さい写真を入れていくのではなく、全面が写真の年賀状もおしゃれですよね。
続きを読む論文のPDFから図を自動で抽出する
この記事は、CAMPHOR- Advent Calendar 2020 の2日目の記事です。
論文のPDFから図を自動で抽出する
こんにちは、シバニャンです。最近やっていることと言えば、修士論文の執筆です。修士論文を書くためには、普段から大量の論文を読んでおく必要があります。僕の研究分野はコンピュータビジョンという画像を中心に扱う分野で、年数回、国際会議があるたびに1000本程の論文が発表されます。その中から自分の研究に関係があったり、直接関係はなくても役立ちそうなアイディアがある論文を見つける必要があります。
論文ギャラリーを作りたい
